SMN(运动神经元存活蛋白)是一种含Tudor结构域的蛋白,主要由2个高度同源的基因SMN1和SMN2编码,其中SMN1的基因缺失导致SMN蛋白的不足,从而导致SMA(脊髓性肌萎缩症)——一种常染色体遗传疾病,其发病率为1/6000~1/10000,是导致新生儿死亡的主要遗传疾病之一。尽管SMN2基因也可以编码表达少量的SMN蛋白,但与SMN1基因相比,SMN2基因由于第7个外显子的第6位存在从C到T的转变导致外显子剪接沉默子位点的产生,导致第7个外显子在mRNA水平跳跃,从而编码表达一个截短的SMN蛋白(图1)。
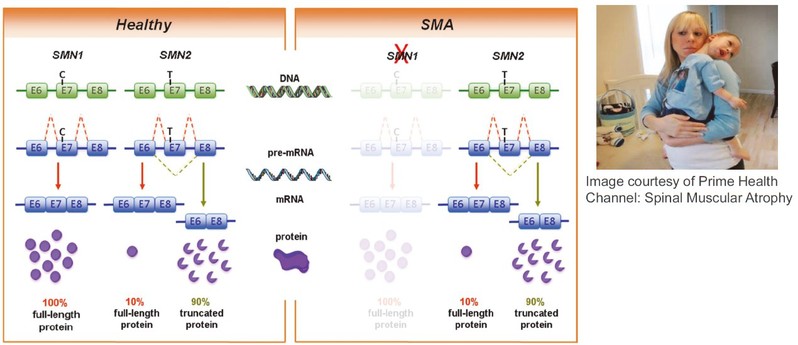
图1 SMN(运动神经元存活蛋白)与SMA(脊髓性肌萎缩症)
生物学功能研究发现,SMN蛋白作为SMN复合物的核心,通过其Tudor结构域识别对称二甲基化修饰的精氨酸(Rme2s)来介导SMN功能的发挥,如SMN通过Tudor结构域与RNA聚合酶II(RNAP II)的POLR2A亚基1810位的Rme2s(R1810me2s-POLR2A)相互作用而调控转录的终止。
尽管对SMN及其相关的SMA疾病已经开展了广泛的研究,但SMN如何保护脊髓运动神经元免受退化的影响仍不清楚。高亲和力、高特异性小分子探针在不改变蛋白表达情况下,是研究蛋白功能的一个有效手段。
为此,苏州大学药学院刘艳丽教授课题组与多伦多大学闵金荣教授课题组合作,利用高通量筛选,获得了SMN Tudor结构域与Rme2s作用界面的小分子拮抗剂(图2a-b),并解析了复合物结构,结果提示小分子拮抗剂占据了Rme2s的结合位点(图2c)。进一步的分子靶向性研究表明,该小分子拮抗剂在细胞内特异性结合SMN(图2d-e)。小分子拮抗剂生物学功能分析表明,拮抗剂破坏了SMN与R1810me2s-POLR2A的相互作用(图2f),减少了SMN在靶基因位点的富集(图2g-h),从而导致RNAP II和R-loop在靶基因位点的过度积累(图2i-j),模拟了SMN基因敲除的结果。因此,除了RNAi和CRISPR/Cas9技术,该有效的SMN拮抗剂可作为研究SMN生物学及其相关神经疾病的有效工具。相关的研究结果以“A small molecule antagonist of SMN disrupts the interaction between SMN and RNAP II”为题,近期发表于Nature communications,2022,13:5453,PMID: 36114190,苏州大学药学院刘艳丽教授是该文章的第一兼共同通讯作者。
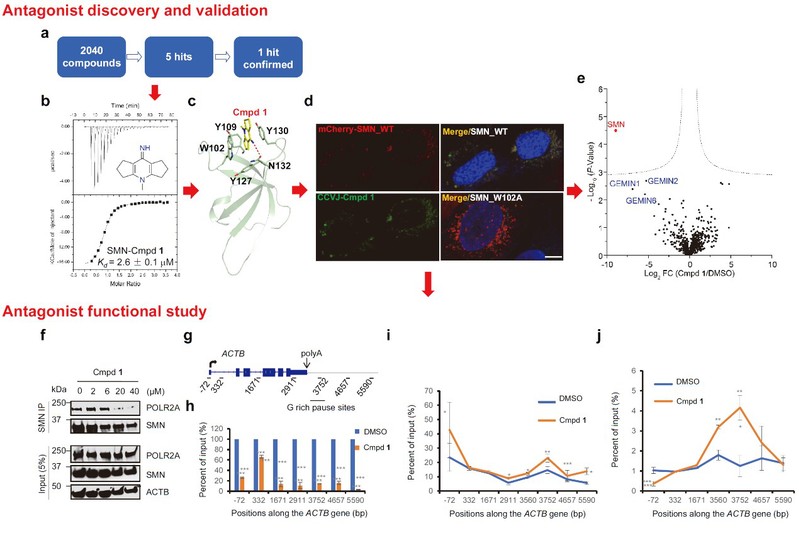
图2 SMN小分子拮抗剂的发现及其生物学功能分析

刘艳丽,苏州大学药学院特聘教授,江苏省重大神经精神疾病研究重点实验室PI。主要从事表观遗传调控相关生物大分子结构和功能研究及基于分子结构的药物研发工作。先后入选湖北省“青年科技晨光计划”和江苏省“六大人才高峰”高层次人才选拔培养资助计划。主持国家自然科学基金2项,以第一作者/共同第一作者/通讯作者在Nat Commun(3篇)、Nat Chem Biol(1篇)、Cell Chem Biol(1篇)、J Med Chem(1篇)、Pharmacol Ther(1篇)、J Biol Chem(3篇)等国际主流期刊发表论文24篇。

